Domesticada hace 6.000 a 10.000 años en el altiplano andino del sur de Perú [1, 2], la papa cultivada (Solanum tuberosum L.) se ha convertido rápidamente en uno de los principales alimentos básicos del mundo [3]. Con una producción global de 377 millones de toneladas en 2016, por un valor bruto total de 111 mil millones de dólares, la papa ocupa ahora el cuarto lugar entre los cultivos alimentarios, detrás del arroz, del trigo y del maíz [4].
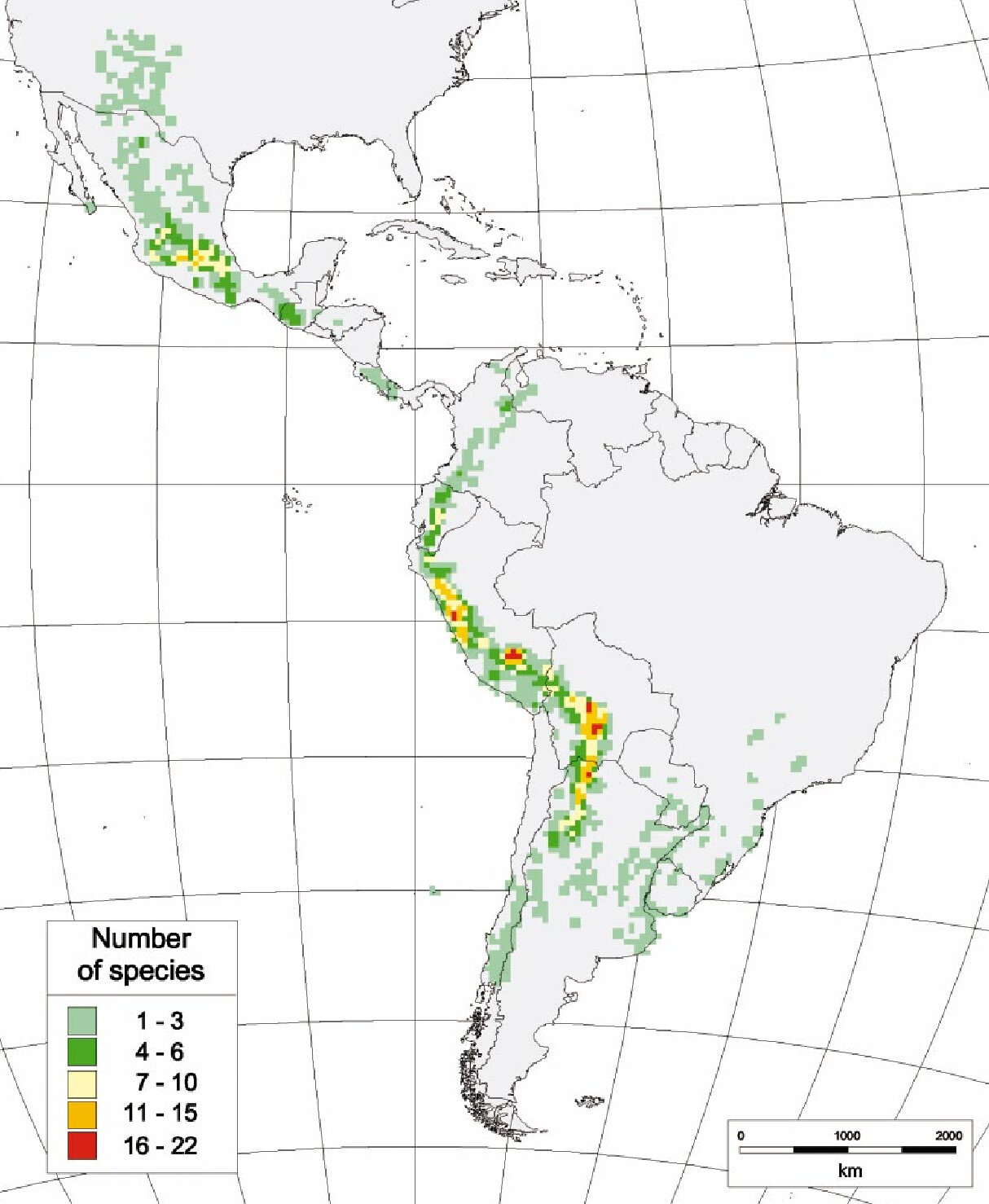
Los esfuerzos actuales de mejoramiento de la papa están dirigidos a aumentar los rendimientos, la compatibilidad con las necesidades de la industria agroalimentaria, la resistencia a los estreses abióticos (por ejemplo, la sequía) y la resistencia a los patógenos [5, 6]. En su búsqueda de rasgos de interés para S. tuberosum, los mejoradores se pueden aprovechar en la gran diversidad botánica de las papas, que incluye 4 especies cultivadas y 107 especies silvestres [2], distribuidas por toda América Latina (Figura 1), desde Nuevo México hasta Patagonia [7, 8].
Al estar adaptadas a una amplia gama de condiciones ambientales y climáticas, las papas silvestres poseen características agrícolas deseables, como una mayor resistencia a las enfermedades y la sequía. Recientes estudios genómicos en el clado de la papa (Solanum sect. Petota) revelaron la extraordinaria diversidad genética de las especies silvestres y su utilidad para la mejora varietal [9, 10].
S. chacoense: un recurso para la mejora de la papa…
Mi proyecto de investigación trata de la biología reproductiva de Solanum chacoense (Figura 2), una especie de papa silvestre cuyo genoma ha sido recientemente secuenciado [11]. S. chacoense se estudia por su alta resistencia a una amplia gama de patógenos, incluyendo el virus Y de la papa [12, 13], las bacterias que causan la marchitez de la papa [14, 15] y la roña común [16, 17], o los hongos responsables de la costra negra y la sarna común [18], y el mildiu [19].
Solanum chacoense también tiene un metabolismo secundario peculiar que produce altos niveles de compuestos glicoalcaloides especiales, tales como la leptina y las leptininas [20], que son particularmente eficaces contra el escarabajo de la papa [21]. Este pequeño insecto herbívoro (Figura 3) es una plaga peligrosa, que puede destruir completamente un cultivo de papa alimentándose de las hojas. Actualmente se trabaja para comprender mejor los genes implicados en estas vías metabólicas [22, 23] y para introducirlos en la papa cultivada [24, 25, 26, 27].
…y una planta modelo para el estudio de la reproducción sexual
Siendo una especie diploide, S. chacoense también se utiliza como un modelo alternativo a la papa cultivada (tetraploide) para la biología molecular y celular, especialmente en el estudio de la reproducción de plantas. Por ejemplo, nuestro instituto de investigación, S. chacoense fue utilizado en el laboratorio Cappadocia para descifrar los mecanismos que gobiernan la autoincompatibilidad gametofitica basada en S-RNasas [28, 29, 30, 31]. El laboratorio Geitmann, ahora basado en la Universidad McGill, también usó S. chacoense en sus investigaciones sobre la citomecánica del crecimiento del tubo polínico [32, 33, 34].
El laboratorio Matton descubrió varias proteínas implicadas en vías de transducción de señales reproductivos en S. chacoense, tales como péptidos RALF [35, 36], MAP quinasas (missing reference) y receptores quinasas [37, 38, 39, 40]. Nuestro grupo también produjo estudios transcriptómicos [41], proteómicos [42] y secretómicos [43] a gran escala para comprender mejor la reproducción de S. chacoense.
Mi proyecto de tesis doctoral tiene como objetivo comprender cómo las interacciones polen-pistilo, especialmente la atracción del tubo polínico, están involucradas en el aislamiento reproductivo de las papas silvestres, es decir, en los mecanismos que les permiten evitar la hibridación interespecífica.


Referencias
- Spooner DM, McLean K, Ramsay G, Waugh R and Bryan GJ. (2005). A single domestication for potato based on multilocus amplified fragment length polymorphism genotyping. Proc. Natl. Acad. Sci. U. S. A., 102(41): 14694–9. DOI: 10.1073/pnas.0507400102
- Spooner DM, Ghislain M, Simon R, Jansky SH and Gavrilenko T. (2014). Systematics, diversity, genetics, and evolution of wild and cultivated potatoes. Bot. Rev., 80(4): 283–383. DOI: 10.1007/s12229-014-9146-y
- Birch PRJ, Bryan G, Fenton B, Gilroy EM, Hein I, Jones JT, … Toth IK. (2012). Crops that feed the world 8: Potato: are the trends of increased global production sustainable? Food Sec., 4(4): 477–508. DOI: 10.1007/s12571-012-0220-1
- Food and of the United Nations AO. (2016). FAO Statistics Division data for year 2016. URL: http://www.fao.org/faostat/en (accessed August 1, 2018).
- Halterman D, Guenthner J, Collinge S, Butler N and Douches D. (2015). Biotech potatoes in the 21st century: 20 years since the first biotech potato. Am. J. Potato Res., 93(1): 1–20. DOI: 10.1007/s12230-015-9485-1
- Hameed A, Zaidi SS-E-A, Shakir S and Mansoor S. (2018). Applications of new breeding technologies for potato improvement. Front. Plant Sci., 9: 925. DOI: 10.3389/fpls.2018.00925
- Hijmans RJ and Spooner DM. (2001). Geographic distribution of wild potato species. Am. J. Bot., 88(11): 2101–12.
- Hijmans RJ, Spooner DM, Salas AR, Guarino L and de la Cruz J. (2002). Atlas of wild potatoes. Rome, Italy: International Plant Genetic Resources Institute.
- Hardigan MA, Laimbeer FPE, Newton L, Crisovan E, Hamilton JP, Vaillancourt B, … Buell CR. (2017). Genome diversity of tuber-bearing Solanum uncovers complex evolutionary history and targets of domestication in the cultivated potato. Proc. Natl. Acad. Sci. U. S. A. DOI: 10.1073/pnas.1714380114
- Li Y, Colleoni C, Zhang J, Liang Q, Hu Y, Ruess H, … Huang B. (2018). Genomic analyses yield markers for identifying agronomically important genes in potato. Mol. Plant, 11(3): 473–84. DOI: 10.1016/j.molp.2018.01.009
- Leisner CP, Hamilton JP, Crisovan E, Manrique-Carpintero NC, Marand AP, Newton L, … Buell CR. (2018). Genome sequence of M6, a diploid inbred clone of the high-glycoalkaloid-producing tuber-bearing potato species Solanum chacoense, reveals residual heterozygosity. Plant J., 94(3): 562–70. DOI: 10.1111/tpj.13857
- Bravo-Almonacid F, Rudoy V, Welin B, Segretin ME, Bedogni MC, Stolowicz F, … Mentaberry A. (2012). Field testing, gene flow assessment and pre-commercial studies on transgenic Solanum tuberosum spp. tuberosum (cv. Spunta) selected for PVY resistance in Argentina. Transgenic Res., 21(5): 967–82. DOI: 10.1007/s11248-011-9584-9
- Duan H, Richael C and Rommens CM. (2012). Overexpression of the wild potato eIF4E-1 variant Eva1 elicits Potato virus Y resistance in plants silenced for native eIF4E-1. Transgenic Res., 21(5): 929–38. DOI: 10.1007/s11248-011-9576-9
- Bae J, Halterman D and Jansky S. (2008). Development of a molecular marker associated with Verticillium wilt resistance in diploid interspecific potato hybrids. Mol. Breed., 22(1): 61–9. DOI: 10.1007/s11032-008-9156-8
- Chen L, Guo X, Xie C, He L, Cai X, Tian L, … Liu J. (2013). Nuclear and cytoplasmic genome components of Solanum tuberosum + S. chacoense somatic hybrids and three SSR alleles related to bacterial wilt resistance. Theor. Appl. Genet., 126(7): 1861–72. DOI: 10.1007/s00122-013-2098-5
- Braun SR, Endelman JB, Haynes KG and Jansky SH. (2017). Quantitative trait loci for resistance to common scab and cold-induced sweetening in diploid potato. Plant Genome, 10(3). DOI: 10.3835/plantgenome2016.10.0110
- Jansky S, Douches D and Haynes K. (2017). Transmission of scab resistance to tetraploid potato via unilateral sexual polyploidization. Am. J. Potato Res., 95(3): 272–7. DOI: 10.1007/s12230-017-9628-7
- Almasia NI, Bazzini AA, Hopp HE and Vazquez-Rovere C. (2008). Overexpression of snakin-1 gene enhances resistance to Rhizoctonia solani and Erwinia carotovora in transgenic potato plants. Mol. Plant Pathol., 9(3): 329–38. DOI: 10.1111/j.1364-3703.2008.00469.x
- Faccio P, Vazquez-Rovere C, Hopp E, González G, Décima-Oneto C, Favret E, … Franzone P. (2011). Increased tolerance to wheat powdery mildew by heterologous constitutive expression of the Solanum chacoense. Czech J. Genet. Plant Breed., 47(Special issue): S135–41.
- Mweetwa AM, Hunter D, Poe R, Harich KC, Ginzberg I, Veilleux RE and Tokuhisa JG. (2012). Steroidal glycoalkaloids in Solanum chacoense. Phytochemistry, 75: 32–40. DOI: 10.1016/j.phytochem.2011.12.003
- Lorenzen JH, Balbyshev NF, Lafta AM, Casper H, Tian X and Sagredo B. (2001). Resistant potato selections contain leptine and inhibit development of the Colorado potato beetle (Coleoptera: Chrysomelidae). J. Econ. Entomol., 94(5): 1260–7.
- Manrique-Carpintero NC, Tokuhisa JG, Ginzberg I, Holliday JA and Veilleux RE. (2013). Sequence diversity in coding regions of candidate genes in the glycoalkaloid biosynthetic pathway of wild potato species. G3: Genes, Genomes, Genet., 3(9): 1467–79. DOI: 10.1534/g3.113.007146
- Manrique-Carpintero NC, Tokuhisa JG, Ginzberg I and Veilleux RE. (2014). Allelic variation in genes contributing to glycoalkaloid biosynthesis in a diploid interspecific population of potato. Theor. Appl. Genet., 127(2): 391–405. DOI: 10.1007/s00122-013-2226-2
- Cooper SG, Douches DS and Grafius EJ. (2009). Combining engineered resistance, avidin, and natural resistance derived from Solanum chacoense bitter to control Colorado potato beetle (Coleoptera: Chrysomelidae). J. Econ. Entomol., 102(3): 1270–80.
- Ginzberg I, Thippeswamy M, Fogelman E, Demirel U, Mweetwa AM, Tokuhisa J and Veilleux RE. (2012). Induction of potato steroidal glycoalkaloid biosynthetic pathway by overexpression of cDNA encoding primary metabolism HMG-CoA reductase and squalene synthase. Planta, 235(6): 1341–53. DOI: 10.1007/s00425-011-1578-6
- Molnár I, Besenyei E, Thieme R, Thieme T, Aurori A, Baricz A, … Rakosy-Tican E. (2017). Mismatch repair deficiency increases the transfer of antibiosis and antixenosis properties against Colorado potato beetle in somatic hybrids of Solanum tuberosum + S. chacoense. Pest Manag. Sci., 73(7): 1428–37. DOI: 10.1002/ps.4473
- Crossley MS, Schoville SD, Haagenson DM and Jansky SH. (2018). Plant resistance to Colorado potato beetle (Coleoptera: Chrysomelidae) in diploid F2 families derived from crosses between cultivated and wild potato. J. Econ. Entomol. DOI: 10.1093/jee/toy120
- Boivin N, Morse D and Cappadocia M. (2014). Degradation of S-RNase in compatible pollen tubes of Solanum chacoense inferred by immunogold labeling. J. Cell Sci., 127(Pt 19): 4123–7. DOI: 10.1242/jcs.154823
- Soulard J, Boivin N, Morse D and Cappadocia M. (2014). eEF1A is an S-RNase binding factor in self-incompatible Solanum chacoense. PLoS One, 9(2): e90206. DOI: 10.1371/journal.pone.0090206
- Soulard J, Qin X, Boivin N, Morse D and Cappadocia M. (2013). A new dual-specific incompatibility allele revealed by absence of glycosylation in the conserved C2 site of a Solanum chacoense S-RNase. J. Exp. Bot., 64(7): 1995–2003. DOI: 10.1093/jxb/ert059
- Liu B, Boivin N, Morse D and Cappadocia M. (2012). A time course of GFP expression and mRNA stability in pollen tubes following compatible and incompatible pollinations in Solanum chacoense. Sex. Plant Reprod., 25(3): 205–13. DOI: 10.1007/s00497-012-0192-5
- Aouar L, Chebli Y and Geitmann A. (2010). Morphogenesis of complex plant cell shapes: the mechanical role of crystalline cellulose in growing pollen tubes. Sex. Plant Reprod., 23(1): 15–27. DOI: 10.1007/s00497-009-0110-7
- Parre E and Geitmann A. (2005). Pectin and the role of the physical properties of the cell wall in pollen tube growth of Solanum chacoense. Planta, 220(4): 582–92. DOI: 10.1007/s00425-004-1368-5
- Parre E and Geitmann A. (2005). More than a leak sealant. The mechanical properties of callose in pollen tubes. Plant Physiol., 137(1): 274–86. DOI: 10.1104/pp.104.050773
- Chevalier E, Loubert-Hudon A and Matton DP. (2013). ScRALF3, a secreted RALF-like peptide involved in cell-cell communication between the sporophyte and the female gametophyte in a solanaceous species. Plant J., 73(6): 1019–33. DOI: 10.1111/tpj.12096
- Germain H, Chevalier E, Caron S and Matton DP. (2005). Characterization of five RALF-like genes from Solanum chacoense provides support for a developmental role in plants. Planta, 220(3): 447–54. DOI: 10.1007/s00425-004-1352-0
- Germain H, Gray-Mitsumune M, Houde J, Benhamman R, Sawasaki T, Endo Y and Matton DP. (2013). The Solanum chacoense ovary receptor kinase 11 (ScORK11) undergoes tissue-dependent transcriptional, translational and post-translational regulation. Plant Physiol. Biochem., 70: 261–8. DOI: 10.1016/j.plaphy.2013.05.036
- Germain H, Gray-Mitsumune M, Lafleur E and Matton DP. (2008). ScORK17, a transmembrane receptor-like kinase predominantly expressed in ovules is involved in seed development. Planta, 228(5): 851–62. DOI: 10.1007/s00425-008-0787-0
- Germain H, Houde J, Gray-Mitsumune M, Sawasaki T, Endo Y, Rivoal J and Matton DP. (2007). Characterization of ScORK28, a transmembrane functional protein receptor kinase predominantly expressed in ovaries from the wild potato species Solanum chacoense. FEBS Lett., 581(26): 5137–42. DOI: 10.1016/j.febslet.2007.10.001
- Germain H, Rudd S, Zotti C, Caron S, O’Brien M, Chantha S-C, … Matton DP. (2005). A 6374 unigene set corresponding to low abundance transcripts expressed following fertilization in Solanum chacoense Bitt, and characterization of 30 receptor-like kinases. Plant Mol. Biol., 59(3): 515–32. DOI: 10.1007/s11103-005-0536-8
- Tebbji F, Nantel A and Matton DP. (2010). Transcription profiling of fertilization and early seed development events in a solanaceous species using a 7.7 K cDNA microarray from Solanum chacoense ovules. BMC Plant Biol., 10: 174. DOI: 10.1186/1471-2229-10-174
- Vyetrogon K, Tebbji F, Olson DJH, Ross ARS and Matton DP. (2007). A comparative proteome and phosphoproteome analysis of differentially regulated proteins during fertilization in the self-incompatible species Solanum chacoense Bitt. Proteomics, 7(2): 232–47. DOI: 10.1002/pmic.200600399
- Liu Y, Joly V, Dorion S, Rivoal J and Matton DP. (2015). The plant ovule secretome: a different view toward pollen–pistil interactions. J. Proteome Res., 14(11): 4763–75. DOI: 10.1021/acs.jproteome.5b00618